Секвенирование ДНК А. Библиотеки генов
В генной инженерии часто требуется выделить отдельный, пока еще не охарактеризованный фрагмент ДНК (DNA), например, с целью определения его полной нуклеотидной последовательности. Такая задача решается путем создании библиотек кДНК. Библиотека ДНК состоит из большого количества векторных молекул ДНК, содержащих различные фрагменты чужеродной ДНК. Например, возможно получить все молекулы мРНК клетки в виде фрагментов ДНК - кДНК (англ. complementary DNA, cDNA) — и произвольно внедрить эти копии в векторные молекулы. Библиотека генов может быть получена путем расщепления ДНК клетки на небольшие фрагменты рестриктазами и последующего встраивания этих фрагментов в векторную ДНК. В качестве векторов для получения библиотек ДНК используются бактериофаги (сокращенно: фаги). Фаги — это вирусы, которые инфицируют бактерии, где их геном реплицируется вместе с бактериальным геномом. Библиотеки генов удобны тем, что в них легко вести поиск необходимых в данный момент фрагментов. Работу начинают с того, что небольшую порцию ДНК, составляющую библиотеку (105-106 фагов), разбавляют, смешивая с клетками бактерии-хозяина, и помещают на питательную среду. Бактерии определенное время растут, образуя на питательной среде плотный мутный слой клеток. При этом бактерии, зараженные фагом, растут медленнее, чем неинфицированные клетки. Это приводит к образованию прозрачных кольцевых зон, «бляшек». Клетки в «бляшке» содержат потомство фагов из первоначальной библиотеки. Образовавшиеся колонии переносят на нитроцеллюлозные или найлоновые фильтры, накладывая их на поверхность слоя в чашке, и слегка подогревают. Если теперь инкубировать фильтр с меченым олигонуклеотидным зондом, соответствующим нуклеотидной последовательности целевого фрагмента, произойдет гибридизация зонда с гомологичной последовательностью ДНК. Место связывания зонда можно определить по радиоактивной или иной метке. После этого выделяют фаг из положительных (несущих метку) бляшек и размножают обычным образом. Большие количества необходимого фрагмента получают с помощью расщепления ДНК рестриктазами.
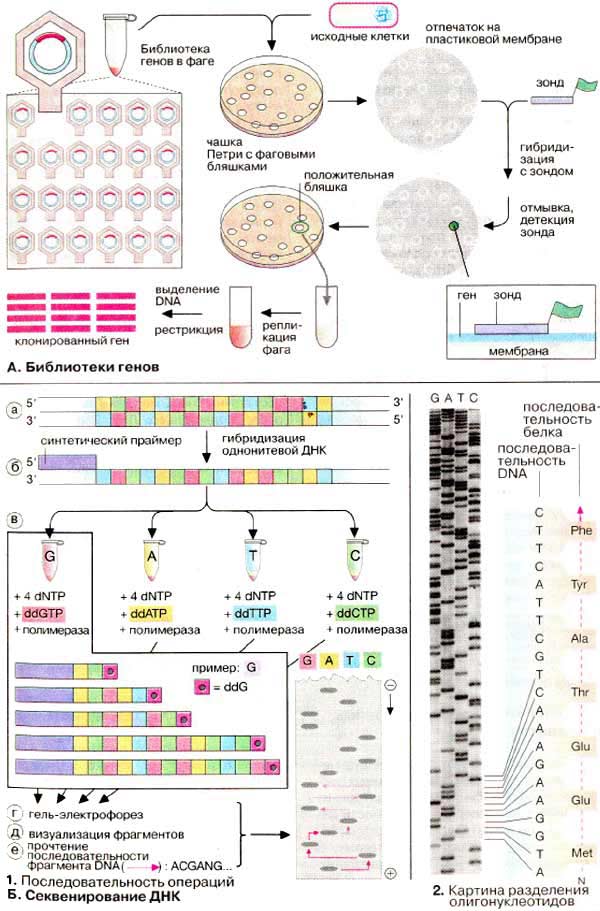
Б. Секвенирование ДНК
Современным методом определения нуклеотидной последовательности ДНК является метод обрыва цепи (англ. chain termination method). Прежде всего фрагмент ДНК клонируют в фаге М13, из которого легко выделяют однонитевую ДНК (а). Ее гибридизуют с короткой ДНК, называемой праймером, которая связывается с 3'-концом однонитевой ДНК (б). Затем к полученной матрице добавляют четыре дезоксирибонуклеозидтрифосфата дНТФ (dNTP): дАТФ (dATP), дГТФ (dGTP), дТТФ (dTTP) и дЦТФ (dCTP). Кроме дезоксирибонуклеозидтрифосфатов в реакционную смесь добавляют один из четырех дидезоксирибонуклеозидтрифосфатов [ддНТФ (ddNTP)]. Затем с помощью ДНК-полимеразы ведут синтез второй (комплементарной) цепи ДНК. Остановка синтеза (обрыв цепи) будет происходить всякий раз, когда вместо дНТФ в растущую цепь ДНК будет встраиваться соответствующий ему ддНТФ. На схеме принцип метода демонстрируется на примере с ддГТФ. В этом случае образуются фрагменты, которые включают праймер плюс 3, 6, 8, 13 или 14 других нуклеотидов. Для определения последовательности нуклеотидов необходимо поставить 4 отдельные реакции в присутствии каждого из четырех ддНТФ (в). Затем четыре пробы вносят в соседние лунки пластины геля и проводят гель-электрофорез (г). Длина пробега каждого компонента обратно пропорциональна длине цепи.
Как только фрагменты ДНК визуализированы (д), нуклеотидную последовательность можно прочесть прямо в геле снизу по направлению к старту в соответствие с очередностью, в которой фрагменты располагаются на отдельных «дорожках» (е). Реальная электрофореграмма с четырьмя дорожками приведена на рис. 2. При оптимальных условиях с помощью метода обрыва цепи можно в одном эксперименте определить строение фрагмента, включающего сотни нуклеотидов.


